近日,我校農(nóng)學(xué)院、廣東省植物分子育種重點(diǎn)實(shí)驗(yàn)室張亞鋒副教授在國際知名期刊Plant Physiology(IF2020=8.34,生物1區(qū))發(fā)表了題為“Mitochondrion-encoded circular RNAs are widespread and translatable in plants”的研究論文(論文鏈接:https://doi.org/10.1093/plphys/kiac143)。該研究開發(fā)了1款適合植物線粒體基因組編碼環(huán)狀RNA(mitochondrion-encoded circRNA,mcircRNA)的識別算法(算法鏈接:https://www.github.com/zyfscau/MeCi),并證明mcircRNA不僅廣泛存在于植物中,而且可以翻譯成蛋白質(zhì)。

近年來,circRNA是非編碼RNA領(lǐng)域的一個(gè)研究熱點(diǎn),相關(guān)研究主要集中在細(xì)胞核基因組編碼circRNA(nucleus-encoded circular RNA,ncircRNA)。ncircRNA廣泛存在于真核生物中,它們主要來自于內(nèi)含子反向剪接途徑。在人類和動(dòng)物中研究結(jié)果表明,ncircRNA可以作為miRNA的海綿體或者被翻譯成蛋白質(zhì)等。目前,常用的circRNA識別算法大多針對ncircRNA設(shè)計(jì)和開發(fā)。采用這些算法,在植物中只鑒定到很少量的mcircRNA,而且它們的功能研究未見報(bào)道。
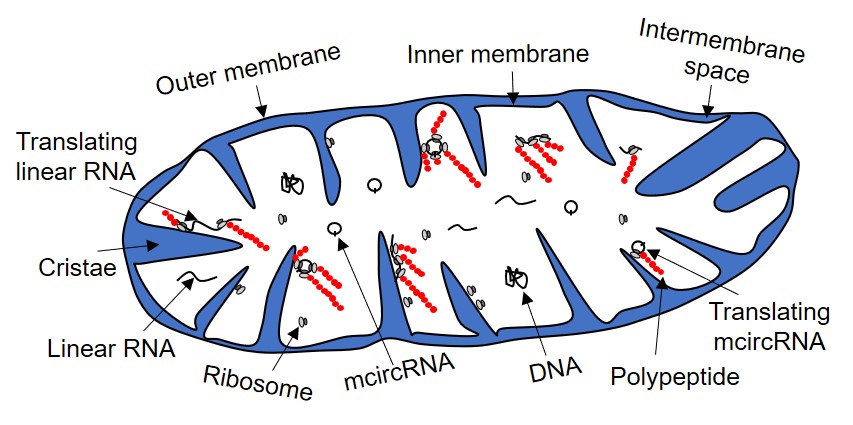
mcircRNA不僅廣泛存在于植物中,并且可以翻譯成蛋白質(zhì)。MeCi算法能夠高效地從轉(zhuǎn)錄組數(shù)據(jù)中鑒定植物mcircRNA
該研究根據(jù)植物線粒體基因組特征(如基因組較小,RNA編輯位點(diǎn)和重復(fù)序列大量存在等)開發(fā)了1款基于blast比對的mcircRNA識別算法,即MeCi。與CIRI2和find_circ常見算法相比,MeCi在mcircRNA預(yù)測數(shù)量、準(zhǔn)確率和計(jì)算效率等方面優(yōu)勢明顯。該研究發(fā)現(xiàn),植物mcircRNA與ncircRNA具有顯著不同的特點(diǎn),它們可能是來源于植物線粒體RNA降解途徑,而不是內(nèi)含子反向剪接。基于線粒體核糖體結(jié)合實(shí)驗(yàn)和質(zhì)譜分析結(jié)果,該研究發(fā)現(xiàn)植物mcircRNA不僅可以和核糖體結(jié)合,并且可以翻譯成蛋白質(zhì)(圖1)。該研究成果豐富了人們對植物circRNA的認(rèn)識,有助于進(jìn)一步揭示植物circRNA的生物合成途徑及其生物學(xué)功能。
我校農(nóng)學(xué)院在讀碩士生廖珣、中國農(nóng)業(yè)科學(xué)院深圳基因組所李小杰副研究員和上海元莘生物醫(yī)藥科技有限公司鄭冠濤博士為論文共同第一作者,張亞鋒副教授為論文通訊作者,廣東省農(nóng)業(yè)科學(xué)院于航博士和農(nóng)學(xué)院黃君博士為論文共同作者。農(nóng)學(xué)院劉向東教授、浙江大學(xué)農(nóng)業(yè)與生物技術(shù)學(xué)院樊龍江教授和褚琴潔博士對該研究工作給予了指導(dǎo)。該研究得到廣東省自然科學(xué)基金面上項(xiàng)目資助。(文圖/農(nóng)學(xué)院)



