近日,,亞熱帶農業(yè)資源保護與利用國家重點實驗室夏瑞團隊在Genome Biology(影響因子10.1)上在線發(fā)表了題為“SynGAP: a synteny-based toolkit for gene structure annotation polishing”的研究論文,。該研究開發(fā)了一種基于基因共線性進行物種基因組基因結構注釋矯正的工具SynGAP(Synteny-based Gene structure Annotation Polisher),。該工具基于近緣物種基因共線性,,去鑒定并矯正原始基因結構注釋中的潛在錯漏,,實現(xiàn)基因結構注釋的優(yōu)化,。

演化過程中,,在具有共同祖先的近緣物種之間,,染色體上同源基因存在保守排列的現(xiàn)象,,被稱為基因共線性(gene synteny),。近緣物種的基因共線性區(qū)塊中,部分基因丟失了與其對應的共線性基因,,進而在區(qū)塊內形成共線性對的間隔(gap,,圖1a)。共線性基因的缺失,,可能由基因組序列的變化引起的,,同時還有可能是錯誤注釋或缺失的基因模型(mis-annotated or absent gene models,MAGs)導致的,?;诤笠环N可能性,作者們通過兩物種的共線性分析,,檢測出共線性區(qū)塊中共線性對的空缺位置(gap區(qū)域),。隨后進行雙向的同源比對以實現(xiàn)對gap內潛在注釋錯漏的初步鑒定與矯正。再通過去冗余,、可靠性指標(R value)計算篩選,、參考注釋質量分級等步驟對初步矯正結果進行質控,最終獲得兩物種的高質量矯正注釋,,并且實現(xiàn)對gap的填補,。通過多個植物、動物物種組合的測試與統(tǒng)計,,明確SynGAP dual可以對被測試基因組GSA進行優(yōu)化——增加優(yōu)質新基因注釋以及共線性基因對,,同時提高了BUSCO完整度。
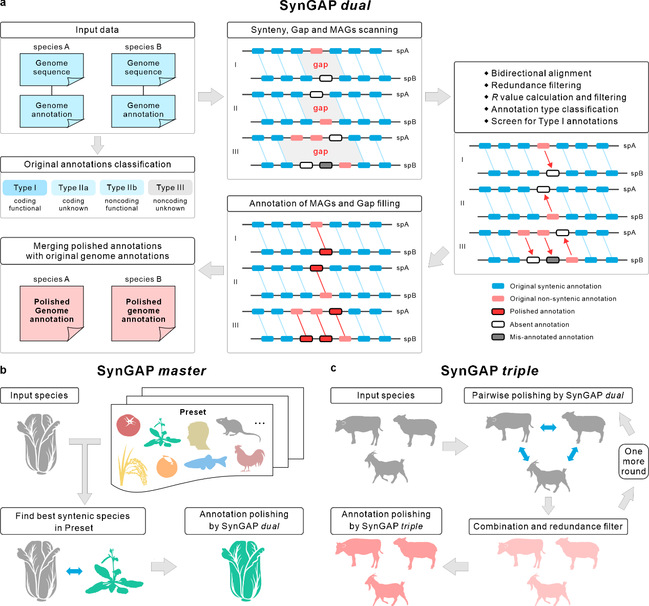
SynGAP基因結構注釋矯正的設計邏輯與流程
除了基因結構注釋矯正功能模塊外,,SynGAP還包含了一套基因物種比較轉錄組分析流程(包含genepair和evi模塊),。通過該流程可實現(xiàn)近緣物種間的準確基因配對,并結合轉錄組數(shù)據(jù)完成跨物種時序性轉錄組分析,高效地篩選鑒定候選關鍵差異表達基因,。其中設計了EVI這一基因差異表達指標,,可同時體現(xiàn)物種間對應基因的表達水平差異、表達量倍數(shù)差異以及表達模式變化差異,?;驅Φ腅VI值越高,兩個同源基因的差異表達就越顯著,。經測試,,EVI可以作為鑒定控制特定性狀或發(fā)育過程(如花色素苷合成、辣椒素合成,、內果皮木質化和大腦體積增大)的候選關鍵基因的有效指標,。
本論文以華南農業(yè)大學為第一完成單位,園藝學院夏瑞教授,、陳程杰博士為共同通訊作者,。博士研究生吳鋒琦為該論文第一作者。博士研究生麥迎曉參與了該論文的軟件功能設計,。該研究得到“十四五”廣東省農業(yè)科技創(chuàng)新十大主攻方向“揭榜掛帥”項目,、國家自然科學基金、廣東省重點研發(fā)項目等資助,。
文圖/亞熱帶國家重點實驗室



